von Felix Günther und Julian Gutberlet 2014
Jana Wins, 2017-01
Die Proteinbiosynthese
Die Proteinbiosynthese (griech. proteos = von äußerster Wichtigkeit) ist der Vorgang in jeder Zelle, der für die Herstellung von Proteinen und Enzymen verantwortlich ist. Die auf diese Weise synthetisierten Proteine bestehen aus Aminosäureketten, deren Länge bis über 1000 Aminosäuren reicht. Die Zusammensetzung, der Aufbau und die Funktion eines Proteins oder Enzyms sind im Erbgut des jeweiligen Lebewesens codiert. Mit Hilfe dieses „Bauplans“ können in Zellen Proteine produziert werden, welche sich in der Abfolge ihrer Aminosäuresequenzen unterscheiden.
Die Genexpression beschreibt, wie die genetische Information eines Gens durch Proteinbiosynthese zum Ausdruck gebracht wird.
Die Proteinbiosynthese lässt sich in zwei Schritte unterteilen. Der erste Schritt ist die Transkription und der zweite die Translation.
(a) Die Transkription: Erstellen einer Kopie der DNA in Form von mRNA und Transport dieser zu den Ribosomen
(b) Die Translation: Übersetzung der genetischen Information der mRNA in die Aminosäuresequenz des Proteins
1 Transkription
Während der Transkription wird eine Kopie eines bestimmten Genabschnitts, welcher auf der DNA liegt, in Form von mRNA synthetisiert.
In der mRNA wird anstatt der Nukleotidbase Thymin Uracil verwendet und die Ribose ist am zweiten Kohlenstoff nicht oxidiert.
Vorteile der Herstellung einer Kopie:
- Die DNA selbst kann nicht aus dem Zellkern transportiert werden, was für den zweiten Schritt der Proteinbiosynthese nötig wäre. Die Information wird transportabel.
- Informationen einzelner Gene können übermittelt werden.
- Gezielte Ausprägung/Aktivierung einzelner Gene ist möglich.
- Durch die Bildung vieler Kopien können mehrere Ribosomen gleichzeitig arbeiten.
- Die DNA kann immer wieder für die Proteinbiosynthese benutzt werden. Die Matritze / Vorlage wird gesichert.
Die mRNA ("Messenger"-RNA) gilt als Bote, welcher die Informationen des bestimmten Gens überliefert. Sie ist eine komplementäre Abschrift eines Gens des codogenen DNA-Stranges. Nur momentan benötigte Informationen der DNA werden "abgeschrieben" (transkribiert), um nicht die ganze DNA kopieren zu müssen.
Die Transkription lässt sich in drei Phasen aufteilen: Initiation, Elongation und Termination.
Initiation:
Zuerst bindet das Enzym RNA-Polymerase an die von ihm erkannte Startstelle (Promotor) auf der DNA. Der Promotor hat eine bestimmte Basensequenz, woran die Polymerase die Startstelle erkennt .
Die DNA wird durch das Lösen der Wasserstoffbrückenbindungen zwischen den Basenpaaren geöffnet. Das Enzym liest nur den codogenen Strang (Matrizenstrang) ab.
Elongation:
Analog zur DNA-Replikation werden nun Nucleosidtriphosphate (ATP, GTP, CTP, UTP) komplementär an den codogenen Strang von der RNA-Polymerase angelagert
(Ablesen: 3‘-5‘-Richtung ; Synthese: 5‘-3‘-Richtung).
Termination:
Durch Terminatorsequenzen in der DNA stoppt die Transkription. Jetzt wird das Transkript (auch Prä-mRNA genannt) von der DNA abgelöst. Im Folgenden löst sich die RNA-Polymerase, woraufhin sich die DNA- Einzelstränge erneut zur Doppelhelix winden.
Da eine identische Abschrift der DNA entstanden ist, wurden auch genleere Anschnitte (Introns) kopiert. Diese werden nun durch eine Prozessierung (Splicing) herausgeschnitten, wodurch die mRNA ausschließlich Exons (Genträger) enthält. Dies ist jedoch nur bei Eukaryoten nötig, da in der Prokaryoten-DNA keine Introns vorkommen.
Transkription
Quelle: Public Domain, http://commons.wikimedia.org/wiki/File:DNA_transcription.svg
Im Detail: Der Begriff „ Transkription“ leitet sich vom lateinischen Verb transcribere ab, was so viel wie um- oder überschreiben heißt.
Zusammengefasst ist die Transkription der Vorgang, die DNA in mRNA ( = messenger- RNA, eine Kopie des codogenen DNA-Strangs) zu umschreiben; danach folgt der Transport der mRNA zum Ribosom, wo die Translation stattfindet. Die Transkription wird in drei Phasen unterteilt: Initiation, Elongation und Termination.
In der Initiation bindet das Enzym RNA-Polymerase an der Startstelle der DNA. Dadurch wird der Promotorbereich zwischen der RNA-Polymerase und der DNA gebildet. Durch das Lösen der Wasserstoffbrückenbindungen zwischen den Basenpaaren öffnet sich der Promotorbereich.
Als nächstes erfolgt in der Elongation die Anlagerung der Nukleosidtriphosphate an den codogenen DNA-Strang. Zu beachten ist hier, dass hier Uracil anstelle von Thymin vorkommt. Die Synthese erfolgt vom 5´ zum 3´ Ende; das Ablesen umgekehrt.
In der Termination wird die Transkription gestoppt, wobei sich die Prä-mRNA, also das neu gebildete Transkript, von der DNA löst. Ebenso löst sich auch die RNA-Polymerase und die DNA-Stränge bilden wieder die Ausgangsform der Doppelhelix.
Bei Eukaryoten wurden auch Introns (= abschnitte ohne Gene) kopiert, die durch Splicing herausgeschnitten werden, damit nur noch Exons ( Abschnitte mit Genen) vorhanden sind.
2 Translation
Drei nebeneinander liegende Basen in der mRNA-Sequnez werden als Triplett (oder Codon) bezeichnet. Sie codieren jeweils für eine bestimmte Aminosäiure. Dies ist der genetische Code.
Da es keine strukturelle Verwandtschaft zwischen dem Codon und der dazugehörigen Aminosäure gibt, wird ein Zwischenstück benötigt, das einerseits die Aminosäure bindet und andererseits das zugehörige Codon auf der mRNA erkennt. Für diesen Vorgang sind Aminosäuretransporter, die tRNAs (Transfer-RNAs) notwendig. Sie besitzen zwei exponierte Bindungsstellen: Das Anticodon und die Aminosäurebindungsstelle (3‘-Ende). Die Aminosäurebindungsstellen der tRNAs werden durch das Enzym Aminoacyl-tRNA-Synthetase spezifisch mit der passenden Aminosäure beladen. Die tRNA erkennt mit dem Anticodon, dessen Basensequenz zum Codon komplementär ist, das entsprechende Codon auf der mRNA und bindet daran.
Auch die Translation lässt sich in diese Phasen aufteilen:
Initiation:
Die mRNA ist nun durch das ER zu einem der vielen Ribosomen (Eukaryoten: 80S, Prokaryoten: 70S) transportiert worden. Diese bestehen aus einer großen (Eukaryoten: 60S, Prokaryoten: 50S) und einer kleinen Untereinheit (Eukaryoten: 40S, Prokaryoten 30S). Zunächst verbindet sich das 3‘-Ende der mRNA mit der kleineren Untereinheit. Diese liest die mRNA so lange ab, bis das Startcodon AUG auftritt. Daraufhin bindet die tRNA mit dem komplementären Anticodon UAC, welches die Aminosäure F-Met bindet, an die P-Stelle. Der sogenannte Initiationskomplex entsteht. Darüber schließt sich die große Untereinheit, wodurch der mRNA-Strang einmal durch das gesamte Ribosom führt.
Elongation:
Eine weitere beladene tRNA tritt in die A-Stelle und bindet mit ihrem Anticodon nach dem Schlüssel-Schloss-Prinzip an das komplementäre Codon der mRNA. Nun findet der Peptidtransfer statt, das heißt, die Aminosäurekette der tRNA in der P-Stelle (F-Met) wird durch eine Peptidbindung auf die Aminosäure der neuen tRNA in der A-Stelle übertragen. Dadurch wird das Polypeptid im Laufe der Elongation immer länger. Jetzt rückt die Peptidyl-tRNA mit der mRNA um ein Triplett in die P-Stelle weiter (Translokation), wodurch die entladenen tRNA in die E-Stelle gelangt und abgegeben wird. Die somit frei gewordene A-Stelle kann erneut besetzt werden und ein weiterer Elongationszyklus beginnt.
Termination:
Wenn nach erfolgter Translokation eines der drei Terminationscodogene (Stoppcodone sind UAA, UAG und UGA, da sie für keine Aminosäure codieren) in der A-Stelle auftritt, wird die Proteinbiosynthese abgebrochen. Die letzte tRNA wird abgespalten und das Polypeptid freigesetzt, welches sofort in die Tertiärstruktur übergeht. Es kommt zur Dissoziation (Molekülzerfall) des Translationskomplexes in die beiden Untereinheiten des Ribosoms.
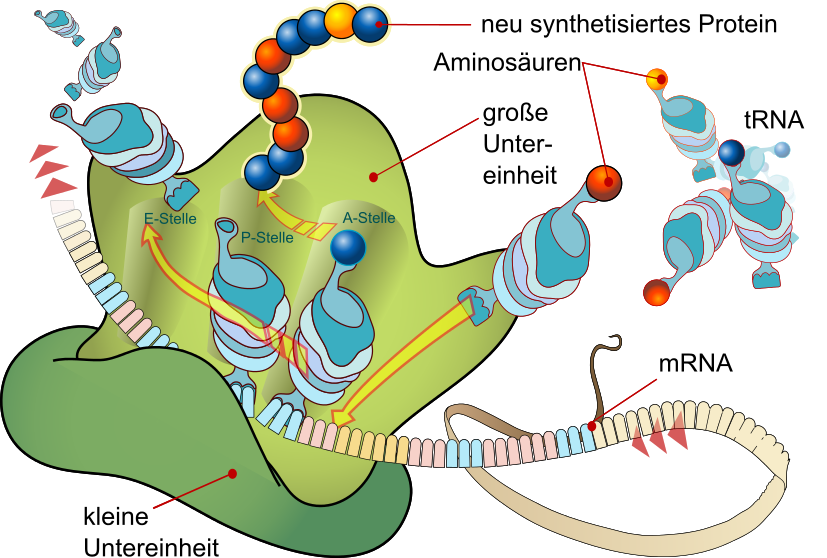
Translation nach dem allosterischen Dreistellenmodell
Quelle: Public domain, Wikicommonsuser: LadyOfhats, Marina Ruiz, http://commons.wikimedia.org/wiki/File:Ribosom_mRNA_translation_de.svg
Im Detail:
Die mRNA kann nun vom Endoplasmatischen Retikulum zum Ribosom, wo schließlich die Translation stattfindet, transportiert werden. Das Ribosom besteht aus einer kleinen und einer großen Untereinheit.
Damit die Aminosäure gebunden und das passende Codon auf der mRNA erkannt werden kann, wird die sogenannte tRNA, also Transfer-RNA, benötigt. Die zwei Bindestellen der tRNA sind das Anticodon, das komplementär zum Codon auf der mRNA ist und so binden kann, und die Aminosäurebindungsstelle.
So wie die Transkription wird auch die Translation in die drei Phasen Initiation, Elongation und Termination eingeteilt.
In der Initiation bindet das 3´-Ende der mRNA an die kleine Untereinheit des Ribosoms. Die mRNA wird bis zum Auftreten des Startcodons AUG abgelesen. Das komplementäre Anticodon zu AUG ist UAC, es bindet die Aminosäure F-Met. Die tRNA mit dem komplementären Anticodon und der Aminosäure bindet an die P- Stelle. Es entsteht der Initiationskomplex, woraufhin sich die große und die kleine Untereinheiten des Ribosoms verbinden und die mRNA einmal durch das Ribosom hindurchgeführt wird. Dies geschieht schrittweise von einem Triplett oder Codon, die sich aus drei nebeneinanderliegenden Basen zusammensetzen, zum nächsten, bis zum Stoppcodon.
In der Elongation tritt eine beladene tRNA an die A-Stelle des Ribosoms. Die Aminosäurekette an der P-Stelle wird jetzt auf die Aminosäure in der A-Stelle übertragen. Dieser Vorgang heißt Peptidtransfer. Sobald dies geschehen ist, rückt die tRNA von der A-Stelle in die P-Stelle des Ribosoms (Translokation), die nun entladene tRNA gelangt in die E-Stelle (Exit- Stelle) und aus dem Ribosom hinausgelangt.
Jetzt ist wieder die A-Stelle frei, die neu besetzt werden kann.
In der Termination wird die Proteinbiosynthese gestoppt, wenn ein Stoppcodon ( UAA, UAG oder UGA) an der A-Stelle angelangt.
Es folgt die Abspaltung der letzten tRNA und zur Freisetzung der Polypeptidkette und das Ribosom zerfällt wieder in die einzelnen Untereinheiten.
Genexpression:
Die Genexpression ist die Ausprägung des Genotyps (genetische Information der DNA) zum Phänotyp eines Lebewesens. Dies wird durch die Proteinbiosynthese ermöglicht.
