Name: Annika Schmitt, 2022-07
DNA-Chip-Technologie
1.1 Einleitung
DNA-Chips oder auch Mikroarrays genannt ermöglichen uns Menschen das Ausmaß der Genexpression in einer Zelle oder einem Organismus zu testen und das menschliche Genom zu entschlüsseln. Sie werden auf kleinen Glas- oder auch Plastikplatten, welche man beispielsweise auch fürs Mikroskopieren nutzt, hergestellt. Diese Platten sind in viele Kästchen unterteilt, in denen kurze DNA-Bruchstücke (DNA- Sonden) aus dem bestimmten Genom geklebt werden. Die einzelnen Felder enthalten also unterschiedliche Basensequenzen, welche einen bestimmten Abschnitt auf der DNA repräsentieren. Diese Basensequenzen jedes Feldes, sowie die Position des Feldes auf dem DNA-Chip werden auf einem Computer gespeichert. Der Chip ist nur ungefähr vier Quadratzentimeter groß, kann aber trotzdem das gesamte menschliche Erbgut beinhalten.
Man unterscheidet im Wesentlichen zwischen zwei DNA-Mikroarrays: Es gibt zum Einen „Spotted Microarrays“, welche ca.50 Nukleotide beinhalten. Diese werden mithilfe von PCR-Produkten auf ein Trägermaterial gedruckt. Die „Oligonucleotide-Microarrays“ (ca. 2.000-3.000 Nukleotide lang) werden mithilfe von künstlich hergestellten Oligonukleotiden synthetisiert. Diese DNA- Fragmente werden auf bestimmte Spots auf das Trägermaterial aufgebracht. Anschließend werden die Gensequenzen, welche für eine spezifische Anwendung gebraucht werden aus einer Gendatenbank ausgesucht.
Der große Vorteil der DNA-Chip- Technologie besteht also darin, dass man für jede Charakterisierung von Zellen, diejenigen Gene betrachten kann, welche von Interesse sind.
Quelle Bild: Public domain by US-National Institutes of Health, NIAMS photograph database & wikicommonsuser Rhoda Baer; https://commons.wikimedia.org/wiki/File:Affymetrix_GeneChip.jpg
2. Herstellung von „Spotted Microarrays“
2.1 Auswahl der gewünschten Bakterienklone
Aus einer cDNA-Datenbank werden die bestimmten Bakterien-Klone ausgewählt, welche die gewünschte DNA-Sequenzen beinhalten. Diese werden dann mithilfe des PCR- Verfahrens repliziert.
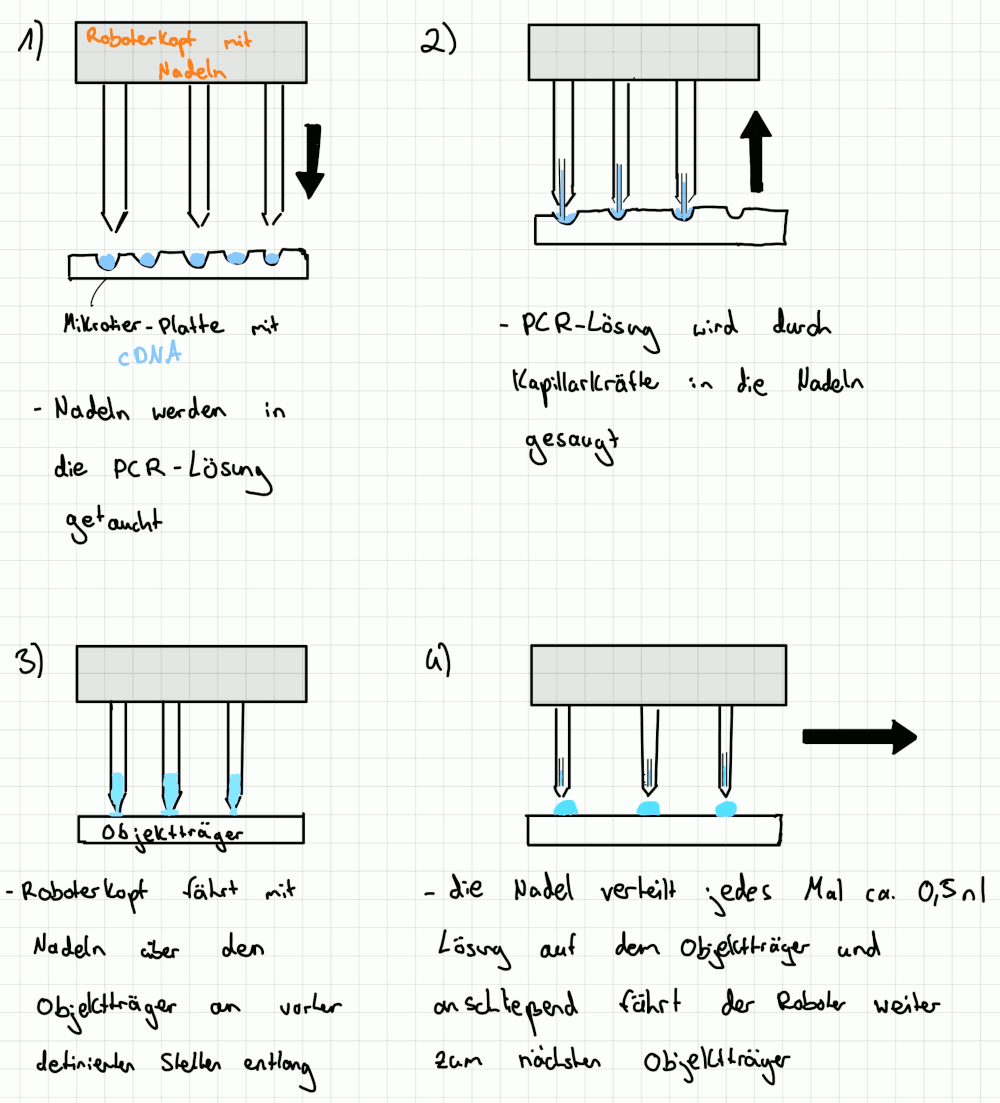
2.2 Aufbringen der cDNA auf den Objektträger (Spotting)
Nachdem die Nadeln über alle Objektträger gefahren sind und die gesamte cDNA aufgenommen worden ist, werden sie ausgewaschen.
2.3 m-RNA-Isolierung
Nun testet man mithilfe der cDNA-Chips ob bestimmte Gene aktiv sind oder exprimiert werden. Dazu isoliert man die mRNA aus einem bestimmten Gewebe. Hierzu kühlt man die Gewebeproben zunächst in flüssigem Stickstoff, da die mRNA sonst sehr schnell zerfällt. Danach müssen die Zellen zerstört werden, um die mRNA vollends zu isolieren, kommt das Gewebe mit einer Metallkugel in ein Gefäß, welches mit hoher Geschwindigkeit geschüttelt wird. Dadurch wird das Gewebe zerstört und anschließend werden bestimmte Chemikalien dazu gegeben, welche die mRNA deaktivieren. Nun kann sie aus dem Gemisch bei Raumtemperatur herausgenommen werden.
2.4 Hybridisierung
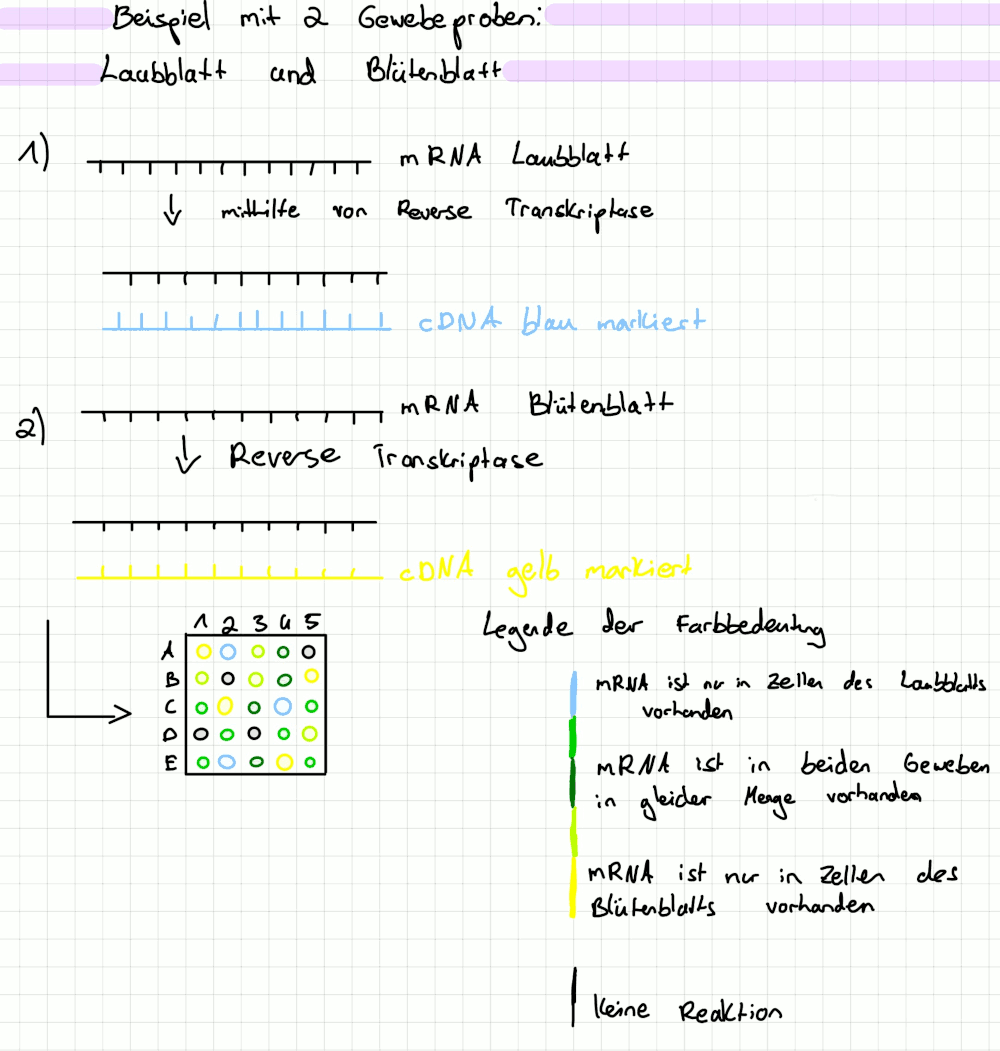
Mithilfe des Enzyms Reverse Transkriptase werden die eher unstabilen RNA-Moleküle in stabile, einzelsträngige cDNA- Moleküle umgeschrieben. Diese werden mit einem bestimmten Fluoreszenzfarbstoff markiert. Die cDNA wird anschließend mit einer Pipette auf die Objektträger mit den Chips aufgetragen und mit einem Deckgläschen abgedeckt, sodass sich die cDNA gleichmäßig verteilt. Über Nacht werden die Objektträger dann in einem Wasserbad bei ca.38 Grad „erwärmt“. Dabei lagern sich die passenden DNA-Abschnitte auf dem Chip an. Dies basiert auf dem Schlüssel-Schloss-Prinzip der komplementären Basenpaarung, denn bei dieser Hybridisierungsreaktion sucht eine DNA-Sonde eine cDNA mit komplementärer Basensequenz heraus, sodass sich dann ein Doppelstrang auf dem Chip bildet. Die cDNA welche sich nicht angelagert hat wird abgewaschen und die Chips können ausgewertet werden.
Quelle Bild: GNU Free Documentation License & Creative Commons Attribution-Share Alike 4.0 International, 3.0 Unported, 2.5 Generic, 2.0 Generic and 1.0 Generic license by wikicommonsuser Magnus Manske - thank you! https://commons.wikimedia.org/wiki/File:Sequenom_chip.jpg
2.5) Auswertung
Die DNA-Chips werden in einen Laser-Scanner gebracht. Der Laser, welcher eine bestimmte Wellenlänge besitzt, erkennt die Fluoreszenz-Farbstoffe auf der cDNA und gibt sie als farbigen Kreis auf dem Computer wieder. Dabei besitzen die Spots mit den meisten gebundenen cDNA- Sequenzen die höchste Farbintensität, welche mithilfe des Scanners eingelesen werden kann. Somit kann die Genaktivität bestimmter Gene festlegen.
Dies funktioniert auch mit zwei verschiedenen Geweben, indem man zwei verschiedene Fluoreszenz-Farbstoffe nutzt, beispielsweise einen, welcher die Farbe Blau und einen der die Farbe Gelb fluoresziert. Anhand der Position, der Intensität und der Farbe kann man überprüfen, ob bestimmte Gene in einem, beiden oder keinem der beiden Gewebe exprimiert werden. Wenn die Sequenz in keinem der beiden Gewebe vorkommt entstehen dunkle/schwarze Spots und wenn sie in beiden Geweben exprimiert werden, entstehen grüne Spots, da blau +gelb -> grün.
3.Anwendung der DNA-Chip-Technologie
Die DNA-Microarrays zählen heutzutage zu den wichtigsten Instrumenten in der Forschung, aber auch die Pharma-Industrie, die Lebensmittel- und Umweltanalytik, sowie die Medizin können einen Nutzen aus dieser Technologie ziehen. Die Pharma-Forschung nutzt sie um bestimmte Wirkstoffe aus einer Substanz herauszufiltern und herauszufinden, was bei der Resistenzentwicklung gegen bestimmte Medikamente auf der genetischen Ebene geschieht.
Es werden beispielsweise Resistenz-Faktoren gegen den Pilz Candida albicans herausgefunden, welcher ein häufiger Auslöser für starke Infektionen im Bereich der Schleimhäute darstellt, welche für Menschen mit einem schwachen Immunsystem durchaus auch tödlich enden können. In der Lebensmittelanalytik dient die Methode dazu, um Lebensmittel, welche gentechnisch verändertes Erbgut oder Keime enthalten, festzustellen. Den größten Nutzen findet die Technologie jedoch wahrscheinlich in der Krebsforschung, um Tumore zu diagnostizieren. Mithilfe der DNA-Chips ist man nun in der Lage mehrere Tausend Gene gleichzeitig mit wenig Aufwand zu analysieren. Dies ist bei dem PCR-Verfahren beispielsweise nicht möglich, da man hierfür nur wenige, ganz bestimmte ausgewählte Gensonden verwendet, welche dann nach Abfolge ihrer Basenpaare analysiert werden.
Die Nutzung von Microarrays ermöglicht eine Klassifizierung von unterschiedlichen Krebsformen und Stadien, denn mithilfe der Chips kann man die Aktivität der Gene von gesunden Zellen mit der Aktivität von Krebszellen vergleichen. Auf diese Weise können die Gene erkannt werden, die bei Tumorzellen besonders aktiv sind. Dies ermöglicht eine potentielle gezielte Krebstherapie, bei der die Wirkstoffe gegen die überaktiven Genbereiche bei der Krebszelle gerichtet sind und sie unschädlich machen.